| このサイトは現在、サイエンス・グラフィックス(株)が管理しています。 お問合せはこちらまで。  |
||||||||||||||||||
| トップページ |
||||||||||||||||||
|
DNAと自ら完成するジグソーパズル、チェス、数学の問題 |
|||||||||||||||||
| 水の中にジグソーパズルのピースとフレームを投げ込んでかき混ぜ、放っておけば完成しているというのは、ありえないことのように聞こえます。しかし、DNAのパズルならこれも可能です。実際、この方法を利用して、DNAチップなどが医療や分析化学に使われています。また、これを利用すればDNAはジグソーパズルを組み立てるだけでなく、難しい数学の問題やチェスまでも解くことができます。 |
||||||||||||||||||
この記事では ・まさか、そのジグソーパズルをそのまま組み立てるおつもりで? ・自己組織化 ・ばらまいたDNAの破片が整然と並んでいく… ・縦に高い従来のコンピュータ、横に広いDNAコンピュータ ・悩める出張セールスマンの旅行計画書とDNAコンピュータ ・山済みにされた問題の中から解決策を見つけられるか という内容で構成しています。 |
||||||||||||||||||
まさか、そのジグソーパズルをそのまま組み立てるおつもりで?
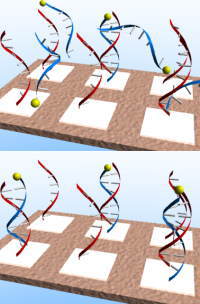
まあ、そのやり方でも悪くはありません。でも、こんな方法はどうでしょう?山済みになっているピースとフレームをまとめて水の中へ放り込み、ゆっくりかき混ぜてあとは待つという方法です。ある程度時間がたてば、ジグソーパズルが組み立てられてしまっているという寸法です。これならあらかじめどんな絵ができるか把握しておく必要もありませんし、まとめていくつかのジグソーパズルを放り込めば、同時にいくつのパズルを組み立てることができます。 もっとも、これは私たちの身近にあるジグソーパズルの話ではありません。そうではなくて、ずっとミクロでピース数の多い複雑なDNAのジグソーパズルの話です。 私たちの感覚からは、なかなかそのイメージがつかめませんが、この方法はDNAチップなどに用いられ、すでに分析化学や医療の現場などでも使われているのです。例えば、ヒトゲノム計画を追い越してしまった民間のセレーラ社のゲノム解析は、これと似た発想を取り入れたものでした。他にも、この方法を取り入れれば、生物兵器のように始めは正体のわからない細菌やウィルスでも、30分程度というわずかな時間である程度正体を特定することができます。 さらには、この発想で、DNAに複雑な数学の問題やチェスを解かせることさえできるのです。DNAが大容量の記憶メディアと同じだということはよく耳にしますが、コンピュータのように情報処理をするということはあまり聞いたことがないかもしれません。しかし、従来のコンピュータとはずいぶん違った方法で、確かにDNAは情報処理をすることができるのです。 そこで今回は、DNAの特異性を積極的に利用して、具体的にどのようなコンピュータをつくろうとしているのかを考えてみることにしましょう。 自己組織化 DNAのジグソーパズルのように、自ら組み上がっていくことを「自己組織化」といいます。この自己組織化は最近のナノテクブームにあわせて急激に注目されるようになってきたのですが、その概念自体は生物学などで古くから知られているものでした。 例えば、いくつかのタンパク質からなるリボゾームは水素結合などでお互いに引き合ってあのような形を保っているのですが、これを洗剤を溶かした水の中に入れてやると、その結合はほどけてバラバラになってしまいます。しかし、洗剤を取り除いてやれば、バラバラだったタンパク質は引き合って再び元のリボゾームの形に戻ります。しかもほとんどの場合、間違って絡まったりすることなく自然に元に戻るのです。これが自己組織化です。 DNAの場合なら、ちょうどよい熱を与えてやることで二重らせん構造が二つのヒモにほどけてしまいますが、普通の温度に戻れば、AとT、CとGが引き合って再び元の形に戻ってしまいます。 こういった比較的単純な自己組織化が繰り返されて、最終的には私たちの目に見える形で、生物という複雑な物体が出来上がっているわけです。生物学では古くから知られた話でしたが、それを積極的に利用して何かをしようと考える人はあまりいませんでした。しかし最近では、ナノテクノロジーなどでこの自己組織化をわずかながら利用できるようになってきたのです。 ばらまいたDNAの破片が整然と並んでいく… 興味深いDNAコンピュータの話に入る前に、仕組みは似ていますが、もう少し単純なDNAチップというものがどうなっているか考えてみましょう。
まずは、あらかじめ配列のわかっているDNA断片(赤)をプレートの上に貼り付けておきます。またプレートのどこにどのDNA断片が貼り付けたかも把握されています。 次に調べたい生物兵器のDNAをバラバラにしていまい、あとで目印となるように蛍光物質(黄)などをつけておきます。そしてこの断片(青)をプレートの上にばらまけば、プレート上のDNA断片とA-T,C-Gという結合による相補性で、DNA断片どうしが自然とペアをつくっていきます。そして、ペアをつくれなかった生物兵器のDNA断片を取り除いてしまえば、プレート上でペアをつくった部分だけが蛍光性を持つことになります。あとはプレートのどの部分が、蛍光物質で光っているかを調べれば、生物兵器が部分的にどのようなDNAをもっているかを知ることができます。これを何度か繰り返すことで、DNA全体がだいたいわかってしまいます。 一見能率が悪そうですが、今では調べたいDNA断片を大量にコピーする技術が確立されているため、上の操作を平行していくつも行うことができ、非常に能率的なのです。 縦に高い従来のコンピュータ、横に広いDNAコンピュータ 先ほどのDNAチップの方法とほぼ同じ発想を利用してつくられたのがDNAコンピュータです。ただしコンピュータといっても従来のものとは全くといっていいほど仕組みが異なります。 これまでのコンピュータのCPUは最初に考え出されて以来、連続的に信号のやり取りを繰り返すというスタイルがずっと守られてきました。この速度が上昇すること以外、あまり他に変化はなかったといえるかもしれません。あのファインマンは、そんなコンピュータの仕組みを「気が狂いそうになるくらい退屈な奴だ」などと言っていたそうです。 一方、DNAの方はどうでしょうか?DNA複製速度が速いといわれる細菌は、一秒間に500程度の塩基対を複製することができるのですが、これをコンピュータと比較しやすくするために言いかえると、1000bits/sec程度となります。DNAコンピュータもずいぶん「退屈な奴」といえるのでしょうか? 確かに、従来のコンピュータと同じ視点から比較している限りは、DNAコンピュータはどうしょうもないくらい役に立ちません。しかしこれでは、DNAコンピュータの本当の魅力は見えてきません。一つのDNAの処理速度は遅いながらも、一つのDNAが複製を完了すれば、DNAが二つになり2000bits/secとなります。次は4つ、8つ…。つまりn回目には2のn乗となり、30回複製したあとでは1000Gbits/secにまでなっているのです。自己組織化するコンピューターです。 まさにこれこそDNAコンピュータの最も優れている点といえます。従来のコンピュータと違って、並列していくつもの処理を行えるというところに利点があるのです。 ではいよいよ、この並列処理によってDNAコンピュータが従来のものと比べてどのようなときに優れた能力を発揮するのかを考えてみましょう。 悩める出張セールスマンの旅行計画書とDNAコンピュータ 現在のCPUにとって苦手な処理といえば、答えの候補が一度にたくさん出てくるものでしょう。その例として古典的な数学の問題に「有向ハミルトン経路問題(directed Hamiltonian Path Problem)」というものがあります。イメージがわきやすいように言いかえれば、「悩める出張セールスマンの旅行計画問題」といったところでしょうか。この悩めるセールスマンの問題を取り上げて、DNAコンピュータがどのような仕組みで情報処理をしているか見てみましょう。 あるセールスマンが飛行機に乗って、セールスのために、いくつかの都市すべてを立ち寄らなければならないとします。ただし、このとき重要なポイントとなるのが、すべての都市どうしがエアラインで結ばれているわけでないということと、一度通った都市は二度と通ってはいけないという制限があるということです。これがセールスマンの悩みの種です。
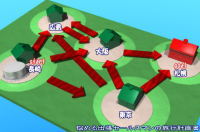
この図の場合、「長崎」から出発して、「広島」、「大阪」、「東京」のすべての都市を一回ずつ立ち寄り、目的地である札幌に向かわなくてはいけません。あなたならどのように旅行計画を立てるでしょうか?それほど難しくありませんね。答えは「長崎>東京>広島>大阪>札幌」という順です。(実際の問題では、都市数がもっと多いが、話しやすくするために5都市にした。) この程度の都市の数なら紙も鉛筆も必要なく、頭の中で簡単に解けるでしょう。しかし、都市の数が増えると、それを結ぶエアラインの数も指数関数的に増えてきます。おそらく都市の数が10を越えるころには、頭の中では解けなくなり、コンピュータに頼ることになります。しかし、今のコンピュータでも1万都市を超えるとなると、多くのハイパワーコンピュータを同時に稼動させても何週間かかかるでしょう。いずれにしても、現在のコンピュータにとってはあまり相性のよい問題ではありません。 この問題は数学的にもコンピュータサイエンス的にもよく出てくる古典的なものなのですが、DNAコンピュータはこれをどのように解くのでしょうか?いくつかのステップに分けて考えてみましょう。 [step1 考えられうるすべての旅行計画を手当たり次第つくる] 何はともあれ、コンピュータが「0/1」の文字に置き換えて処理しているように、DNAコンピュータもDNAの扱いやすい文字、つまり「A,T,C,G」に置き換えてやる必要があります。そこで、5つの都市を区別できるように、次のように置き換えてやりましょう。以下のようなDNA断片をつくります。
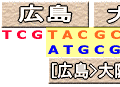
この方法で置き換えれば、旅行経路を、都市の順番(「長崎」>「東京」>「広島」>「大阪」>「札幌」)に並べた'ATGCCGCTACGGTCGTACGCTACGCTAGTA'と、エアラインの順(「長崎>東京」、「東京>広島」、「広島>大阪」、「大阪>札幌」)に並べた、'GGCGATGCCAGCATGCGATGCGAT'が、頭尾の3文字を除いて相補性を保っているという仕組みになっています。
もちろんこれだけで、正しい旅行計画を表すDNA断片だけができるわけではありません。むしろ正しい計画書はほんのわずかで、あとのDNA断片の集まりは数が足りなかったり配列が間違っていたりと、デタラメなものばかりができているはずです。そのため、正しい旅行計画に相当するDNAだけを取り出してやる必要があるのですが、それはまるで大草原の中から小さな藁を拾い上げるようなもので、簡単なものではありません。 そこで、次にどうやって正解の旅行計画を見つけ出すかを考えてみましょう。 [step2 デタラメな旅行計画書の山から、正しいものを取り出すには…] 1.試験管の中には、さまざまの長さや配列のDNA断片が存在しているはずです。そんな中でも、まずは出発点が「長崎」、最終地点が「札幌」になっているもの、つまりDNA断片の端が「長崎」と「札幌」を表す配列になっているものだけを拾い上げたいのです。これは、「ポリメラーゼ連鎖反応(PCR)」で「プライマー(primer)」という相補性を持ったDNA小断片を利用することで可能になります。私たちが欲しいDNA配列は出発点が「長崎(ATGCCG)」で最終地点が「札幌(CTAGTA)」のものです。そこで、それと相補性をもったプライマー「長崎'(TACGGC)」と「札幌'(GATCAT)」を用意します。これによって、端に「長崎」と「札幌」をもつものばかりが複製されます。(ポリメラーゼ連鎖反応(PCR)については下に詳しい解説サイトを紹介)
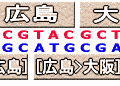
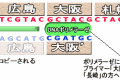
2.次に、上で選択した計画書の中から、5つの都市だけを通っているものだけを選び出しましょう。ここでは、ゲルの上にDNA断片を染み込ませ両側から正負の電気をかけて、負に帯電したDNA断片を泳がせる「電気泳動」という操作を行います。このとき、DNA断片の長さ(重さ)に応じて、泳ぐ距離が変わってくるので、30塩基対(6塩基対×5都市)のものだけを選び出すことができるというわけです。こうして図3のBは取り除かれます。 これでだいぶ計画書を絞り込めましたが、まだデタラメなものが含まれています。例えば「長崎>大阪>広島>大阪>札幌」(図3のC)というような計画書が含まれているはずです。つまり、すべての都市を1度だけずつ立ち寄るという条件を満たしていないものです。そこで、今度はすべての都市に立ち寄っている計画書だけを選ぶために、次のような方法を考えます。 3.たえば、東京(CTACGG)に立ち寄る計画書を拾い上げるために、東京と相補性をもった「東京'(GATGCC)」というDNA断片で、計画書のDNA断片を探します。このとき東京を含んでいない計画書があれば、それは「東京'」とうまくくっつかないので拾い上げられることはありません。つまり、「長崎>大阪>広島>大阪>札幌」という間違った計画書はそのまま見捨てられてしまうのです。こうして、5都市すべてに相補性をもったDNA断片で拾い上げの操作を行います。 この一連の結果得られるものは、出発は長崎、最終は札幌で、5つの都市すべてを5回だけで旅行するというもの・・・つまり、それこそが完璧な旅行計画書なのです(図3のD)。 [step3.完璧な旅行計画書の都市の順番を吟味する] さて、完璧な旅行計画書を選び出すことはできましたが、そのATCGの配列を読んで都市がどのような順番で並んでいるかを知らなくては、旅行計画書を書いたことにはなりません。現在では、直接DNAの塩基配列を読むことのできる特殊な顕微鏡(STMなど)があるのですが、それらを使わない方法を考えてみましょう。 ここではstep2のはじめに行ったプライマーを利用したポリメラーゼ連鎖反応(PCR)を行います。例えば、「大阪」の位置にプライマーを取り付けてPCRを行うとしましょう。すると「長崎、…、大阪」のDNA断片ができます。それの塩基数を数えれば24塩基となるはずです。したがって24÷6=4で大阪が4番目ということになります。
あとは同じようにして、「東京」、「広島」とプライマーをつけてPCRを行えば、12,18塩基となり、2番目、3番目と判断することができます。 これでついに完璧な旅行計画を書き上げることができたのです。出張セールスマンの悩みは解消されました。 山済みにされた問題の中から解決策を見つけられるか これまで長々と話してきたセールスマンの旅行計画書の問題が、実際にDNAを使って実験されたのは、1994年のことでした(このときは20都市で行われた)。 それから5年以上が経ち、ヒトゲノム解析が終わるなど、DNAコンピュータに深く関連している分野は大きく進歩し、それに伴って、ゲノム解析などの操作の多くが機械化されました。例えば、step2,step3のややこしい操作も、今では機械でほぼ自動化することは可能です。 ところが、現時点でもDNAコンピュータがいつ登場するかということになると、誰もはっきりしたことはいえません。5年以上も前にあれほど輝かしい成果があげられたのに対し、今でも問題が山ずみで、DNAコンピュータの実現へはまだ時間がかかるのです。しかし、それはなぜでしょうか?どういった問題があるのでしょうか? まず、小型化が難しいということがあります。94年のセールスマンの実験のときには、試験管の中で行われましたが、この方法では小型化などという言葉と無縁です。もっとも、現在では試験管の中以外でも、金属プレートにDNA断片を固定して実験を行うなどして、一連の操作をしやすくしていますが、やはり小型化には難しい課題が山済みになっています。このままでは高集積のコンピュータをつくることは非常に困難です。 また、DNAの複製などの際には、酵素などの読み違えなどを起こし、エラーが生じることは避けられません。エラーが起こる割合は非常に小さいのですが、ポリメラーゼ連鎖反応のように何度も複製を繰り返していると、エラーの影響がだんだんと大きくなって誤差が無視できなくなるという問題が起こります。これも、DNAコンピュータ実現への大きな障害です。 なにより、DNAコンピュータに本当に向いている処理方法というものが、いまだはっきりしていないことに大きな問題があります。94年のときのセールスマンの計画書に関する実験方法では、他の問題を解くのに応用しにくいということがあります。 そのため、これまでにセールスマンの旅行計画以外にも、チェスの解き方や、ピザのトッピングの仕方など、別のさまざまな方法が試されてきました。しかし、いずれの場合も、DNAコンピューティングのごく限られた一例に過ぎません。DNAのどんな特徴に注目して情報処理に応用するかを見つけ出せるかどうかが、DNAコンピュータ、そして分子コンピュータの成功の大きな鍵と考えられているのです。 ※ポリメラーゼ連鎖反応(PCR)について PCRについては、アニメーションで見るのが一番わかりやすいでしょう。 http://vector.cshl.org/Shockwave/pcranwhole.html |
||||||||||||||||||
| 関連コラム 以前の気になる化学ニュース調査から関連のあるものをあげておきます。 「バイオテロの可能な時代:最先端での攻防」 生物兵器の検出のために、DNAチップなどが使われていることなど。 関連サイト 今回はハミルトン経路を取り扱ったDNAコンピューティングについて書きましたが、実際のところ、この問題がDNAコンピュータに向いているかどうかは難しいところです。ただ、DNAコンピュータのデモンストレーションとして最初に行われたのがこれですし、DNAコンピュータの特徴を説明するのに便利かなと思い取り上げてみました。他にもチェスやピザのトッピングに付いてのものがあるので、いくつか関連サイトを眺めてみてはどうですか。 ・Laboratory for Molecular Science(英語) 94年に世界で一番最初に、ハミルトニアン経路問題を使ってDNAコンピュータのデモンストレーションしたエイドルマンの研究室のホームページ。 ・Interview - Leonard Adleman(英語) エイドルマンへのインタビュー 96年 ・Adleman's Lecture on Molecular Computing - MSRI(英語) エイドルマンの公演のビデオ収録。ページの左上の方をクリック。start 56kps video ・Game of life: Biocomputers take their first step with a little chess puzzle- Princeton(英語) チェスを題材にしたDNAコンピューティングのデモンストレーション ・DNAコンピューティングで重要な成果 - Hotwired DNAコンピューティングを試験管のなかから金属平面上にうつしたときのニュース。 ・ナノコンピューター実現に大きな一歩 - Hotwired ・Time to Engineer DNA Computers - eeTimes(英語) ・The future: biodetectors - Spectrum(英語) 生物兵器の分析に使われるDNAチップなど。 ・DNA Computing: A Primer - arstechnica(英語) DNAコンピューティングについて詳しい解説 ・分子コンピュータプロジェクト - 東大 ・DNAコンピューティング - 北大 |
||||||||||||||||||
| なる_科学ニュース |
||||||||||||||||||