■DNAコンピュータ
− いろいろな酵素をつかった複雑なDNAコンピュータ
エイドルマンの発想は、DNAコンピュータの実現の可能性を示すものだったが、94年にエイドルマンが提案した方法のままでは、実用的なDNAコンピュータを作るのが難しいことはわかっていた。例えばエイドルマンの実験では、正しい答えとなるもの以外にも多くのDNA断片ができてしまう。そのため、あらかじめ必要となるDNAの量は、問題が複雑になるにつれて急激に増えてしまう。また、間違った答えのDNA断片を取り除くための操作回数も同様に増える。このような煩雑な操作や複雑な処理を自動で行なってくれるようなナノマシーンをつくることはできないだろうか?
残念ながら、現在のナノテクノロジーの技術ではこういったナノマシーンを組みたてることはできない。しかし、身近なところにナノマシーンは存在している。生体内では、すでに何億年も前からナノマシーンは存在している。何千、何万種類と存在している酵素を利用すればよいのだ。エイドルマンの94年の報告で使われていた酵素は、DNAリガーゼとDNAポリメラーゼだったが、なにも酵素はこれだけではない。
| 酵素 |
役割 |
| ポリメラーゼ |
DNA鎖の伸長・複製 |
| リガーゼ |
DNA鎖の貼り付け |
| エクソヌクレア−ゼ |
DNAの短縮 |
| 制限酵素 |
DNA特定部位の切断 |
| 修飾酵素 |
DNAの修飾(制限酵素からの保護) |
主なDNA関連酵素と、DNAコンピュータへの利用方法
|
たとえば、ウィスコンシン・マディソン大学の研究チームでは、蛍光体といくつかの酵素を利用することで、エイドルマンの実験では煩雑だった正しい答えの読み出し作業を大幅に簡略化している。[1]
他にも、特定の塩基配列を認識しDNAを切断する「制限酵素」を利用して、複雑な計算処理を行なう実験も行なわれている。このページでは、この制限酵素をうまく使ったイスラエル(Weizmann
Institute of Science)のE.Shapiroらの研究チームについて取り上げてみよう。[2]
一般に制限酵素というのは、例えば、EcoRIのように認識配列の部分を切断することが多い(「DNAコンピュータ/DNA基礎」)。しかしShapiroらの利用した制限酵素FokIは、認識配列からすこし離れた任意の塩基配列(9/13塩基目)を切断するという特殊なものだ。
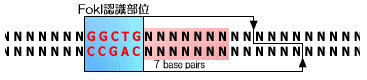
制限酵素FokIのはたらき |
DNA分子による有限制御部分の設計
この研究チームが制限酵素を使った実験で題材にした数学的なモデルは、2つの内部状態(S0,S1)と2つの入力(a,b)をもつ有限オートマトン(finite
automaton)だった。オートマトンというのは、もともとは自動人形という意味だったが、とくにコンピュータサイエンスの世界では、一定の手続きにしたがって情報をある形から別の形に変換する装置という意味で使われている。入力テープを挿入すると、オートマトンはテープにそって動きながらデータを読み取り、一連の計算処理後にはその結果をテープに打ち出す。その模式図を下に示す。
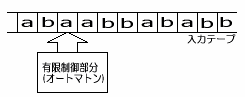
有限オートマトンのモデル図
有限オートマトンは、全体を制御する有限制御部分と、入力記号列が書かれている入力テープとからできていると見なせる。テープヘッドにより左から右に順に1個ずつ入力記号が有限制御部分に読み込まれる。ある状態で記号を読むと新しい状態へ遷移し、次のステップでは新しく遷移した状態で次の入力記号をよんで同様の動作を繰り返す。 |
一般に、2つの状態と2つの入力を持つ有限オートマトンには、以下の8つの遷移規則が存在している。そのため、遷移規則の選択の仕方は2^8-1=255通り存在している。また、最初に信号を受理できる状態は(S0,S1,S0/S1)の3つなので、その選択の仕方は3通り存在している。したがって、最大765(=255X3)通りのプログラムが書けることになる。
| T1 |
S0→S0(a入力) |
| T2 |
S0→S1(a入力) |
| T3 |
S0→S0(b入力) |
| T4 |
S0→S1(b入力) |
| T5 |
S1→S0(a入力) |
| T6 |
S1→S1(a入力) |
| T7 |
S1→S0(b入力) |
| T8 |
S1→S1(b入力) |
遷移規則表;2状態2信号のとき
この765個のプログラムのなかで、面白そうな具体例を一つ取り上げてみよう。
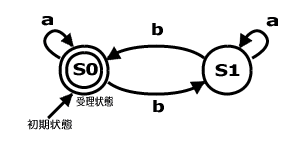
奇偶性検出の有限オートマトン
上の遷移規則表で、T1,T4,T6,T7から構成されている。 |
この図の場合では、bが偶数回でないと入力が受理されない(最終状態が受理状態S0にならない)。つまり、bの数の奇偶を判断できる。つまり、aba,aa,...といった入力は受理されず、abab,aabb,...といった入力が受理されるわけだ。
実際にこのような処理を行なうためには、プログラムの書き込まれた有限制御部分をDNA分子を使って組み立ててやる必要があるが、具体的にどのように行なったかは、以下のサイトなどを参考にしてほしい。
 Biological Nanocomputers: Why and How? - Shapiro Team, Weizmann Institute Biological Nanocomputers: Why and How? - Shapiro Team, Weizmann Institute
 Visual Presentation - Shapiro Team, Weizmann Institute Visual Presentation - Shapiro Team, Weizmann Institute
120mlの液体の入った試験管では、入力分子であるDNA断片が10^12個分だけ、並列して同一の計算を進行している。そのときの全体の1秒間あたりの遷移回数は10^9回程度で、計算の成功率は99.8%程度だったという。また、消費電力はほとんどないに等しい。
さらに、それぞれの入力分子の反応(計算処理)は独立して行なわれている個とを利用して、同一の試験管内で2つの異なるプログラムを同時に行なわせても、期待通りに2つの反応が並列して起こることが確認されている。この実験では、並列処理をはじめとして省スペース、省エネなどDNAコンピュータの将来的な可能性を示すものとなっている。
ref.
[1]Liman Wang, Jeff G. Hall, Manchun Lu,
Qinghua Liu & Lloyd M. Smith, "A
DNA computing readout operation based on
structure-specific cleavage" Nature
Biotechnology November Vol.19 Num.11 pp 1053
(2001)
[2]Benenson, Y. et al. "Programmable
and autonomous computing machine made of
biomolecules" Nature, 414, 430 - 434,
(2001)
|
|